植物次生代谢产物复杂香豆素(complex coumarin)具有多样化的生物活性,如抗菌、抗肿瘤、抗氧化、抗凝血和抗炎等,具备良好的临床应用前景。在自然界中,复杂香豆素仅分布在被子植物的少数谱系—伞形科、芸香科、豆科和桑科。这几个科中,伞形科具有最高的复杂香豆素结构多样性(线性呋喃香豆素、线性吡喃香豆素、角型呋喃香豆素和角型吡喃香豆素),但并非所有伞形科物种都能够合成复杂香豆素,且物种间合成复杂香豆素的含量和类别也存在显著差异。虽然伞形科复杂香豆素生物合成通路已被破译(Zhao et al, 2023; Li et al., 2024),但造成其在伞形科中的分布偏性、类型和含量的多样性差异的机制仍然是个迷。
2024年8月11日,金沙集团3354cm金沙集团3354cm/前沿交叉研究院薛佳宇团队与合作者在《自然·通讯》(Nature Communications)上发表了题为“The gradual establishment of complex coumarin biosynthetic pathway in Apiaceae”的研究论文,重构了伞形科中复杂香豆素生物合成通路起源与进化的详细过程,揭示了形成科内复杂香豆素产物多样性的分子机制。

伞形科复杂香豆素的生物合成涉及3个关键步骤,分别对应3个关键酶基因——香豆酰辅酶A 2’-羟化酶(p-coumaroyl CoA 2’-hydroxylase, C2’H)、异戊烯基转移酶(C-prenyltransferase, C-PT)和环化酶(cyclases)。作者首先系统调研了伞形科不同植物中的香豆素类别和含量情况,然后对这编码三个酶的基因组家族进行分子系统学、比较基因组学和酶活功能的验证,推断C2’H编码基因起源于芹亚科早期,C-PT和环化酶起源较晚,在芹亚科演化后期的滇芹分支(Sinodielsia Clade)、芹族(Apieae),环翅芹族(Tordylieae)、芫荽族(Coriandreae)和亮蛇床族(Selineae)的共同祖先才出现(图1)。
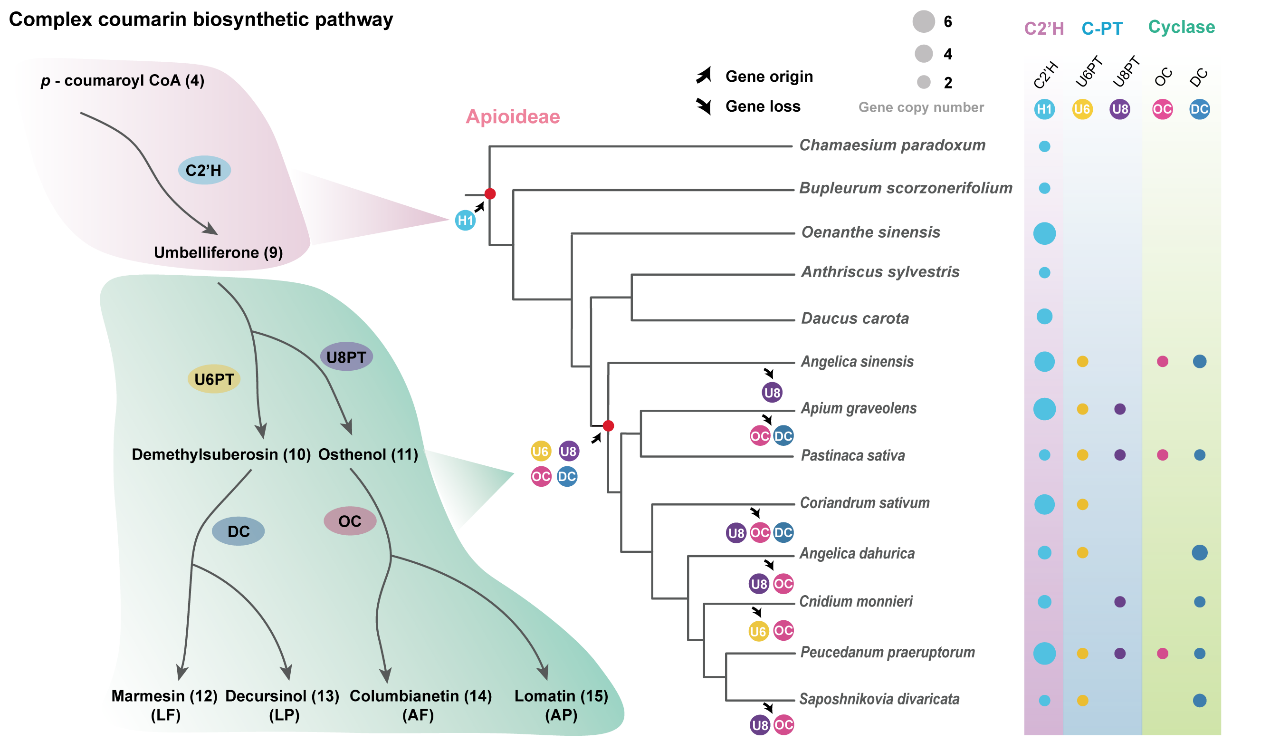
图1. 伞形科复杂香豆素生物合成通路中关键酶基因起源与丢失的总结示意图
伞形科复杂香豆素的产物多样性主要源于C-PT的酶活功能变异,演化出针对于底物伞形酮(Umbelliferone)的不同位置(C-6/C-8)的催化活性。作者通过比较线性和角型构型复杂香豆素合成的C-PT蛋白序列,分析筛选出6个潜在的关键氨基酸位点(图2A),通过定点突变和酶活验证实验发现其中一个氨基酸的变异(Ala161/Thr161)会造成酶活性的改变,在合成线性和角型产物之间来回切换(图2B)。通过酶蛋白三维结构的分析,作者提出该位点通过改变该位置上氨基酸的疏水/亲水性,从而结合底物的不同位置(疏水/亲水面),最终造成产物的线性/角型构型差异(图2C)。
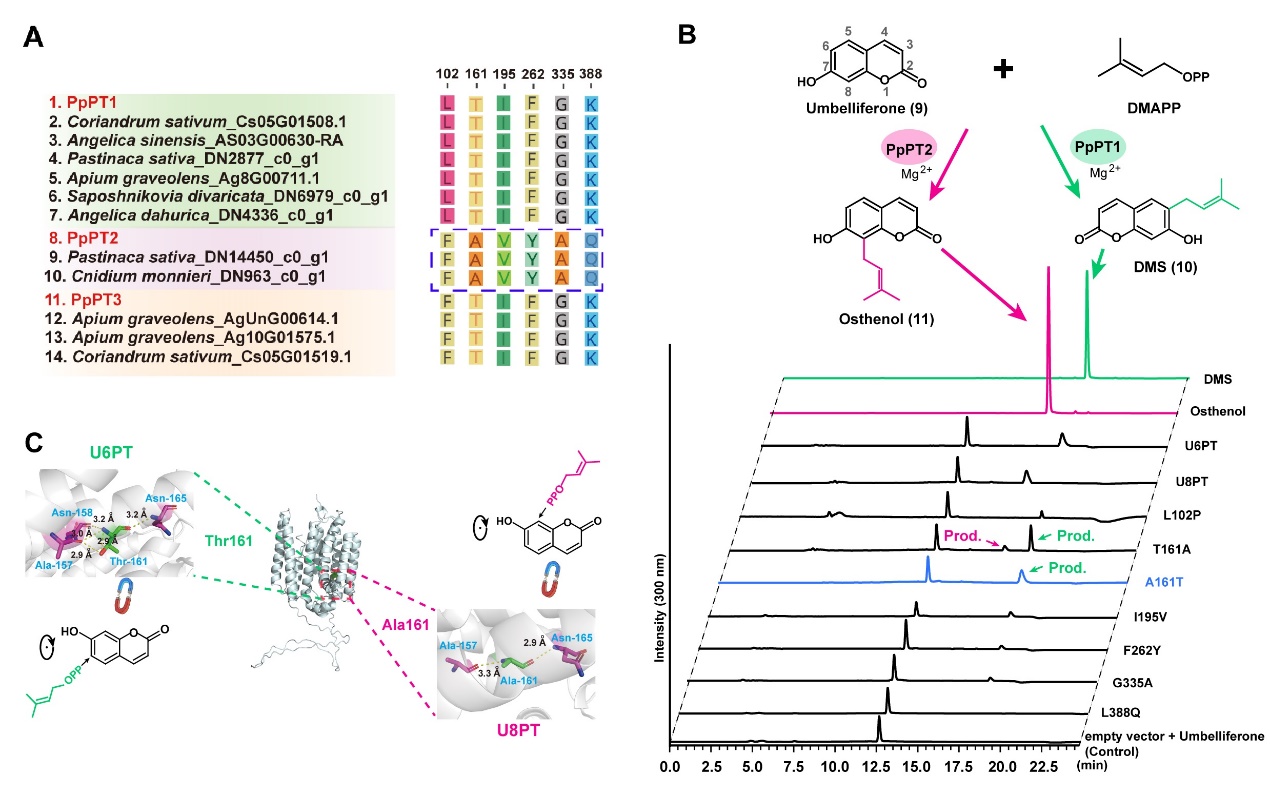
图2. 伞形科复杂决定香豆素线性和角型构型的C-PT蛋白序列分析、酶活实验和作用机制模型
综上,研究人员认为伞形科复杂香豆素合成所需的酶在芹亚科演化后期才最终装配齐全,从而形成完整的生物合成通路,这合理解释了复杂香豆素植物在芹亚科中相对集中的分布模式。完整的合成通路在芹亚科中建成以后,随着后代类群/物种的不断分化,编码这些关键酶的基因在后代谱系中发生多次独立丢失,引发合成通路的部分或完全坍塌,导致有些现有芹亚科物种部分/或完全丢失了合成复杂香豆素的能力,造成了复杂香豆素在芹亚科物种中呈“补丁”式分布的现象以及物种间产物差异(图1)。此外,发现了前胡中形成角型产物的3个关键酶的编码基因的在9号染色体上形成了典型的“生物合成基因簇”(biosynthetic gene cluster)的结构,且明显通过串联复制造成拷贝数量增加,由此解释前胡高效合成积累角型复杂香豆素的内在机制。
金沙集团3354cm为该论文第一和通讯单位,金沙集团3354cm/前沿交叉研究院博士生黄新程、韦雪芬和中国药科大学硕士生唐欢迎为论文共同第一作者,金沙集团3354cm薛佳宇副教授、中国药科大学赵玉成副教授和中国热带农业科学院热带作物遗传资源研究所乔飞研究员为论文共同通讯作者。金沙集团3354cm博士生户帅雅和吴嘉宜、湖南农业大学何岳东博士和陕西中医药大学徐顶巧博士参与了此工作。
论文链接:
https://www.nature.com/articles/s41467-024-51285-x?utm_source=rct_congratemailt&utm_medium=email&utm_campaign=oa_20240810&utm_content=10.1038/s41467-024-51285-x
参考文献:
Yucheng Zhao, Yuedong He, Liangliang Han, Libo Zhang, Yuanzheng Xia, Fucheng Yin, Xiaobing Wang, Deqing Zhao, Sheng Xu, Fei Qiao*, Yibei Xiao*, Lingyi Kong*. 2024. Two types of coumarins-specific enzymes complete the last missing steps in pyran- and furanocoumarins biosynthesis. Acta Pharm Sin B. 14(2):869-880.
Qien Li, Yiqun Dai, Xin-Cheng Huang, Lanlan Sun, Kaixuan Wang, Xiao Guo, Dingqiao Xu, Digao Wan, Latai An, Zixuan Wang, Huanying Tang, Qi Qi, Huihui Zeng, Minjian Qin*, Jia-Yu Xue*, Yucheng Zhao*. 2024.The chromosome-scale assembly of the Notopterygium incisum genome provides insight into the structural diversity of coumarins. Acta Pharm Sin B. 14(8), 3760-3773.
(编辑:薛佳宇 校对:王乙明 审核:陶书田)